
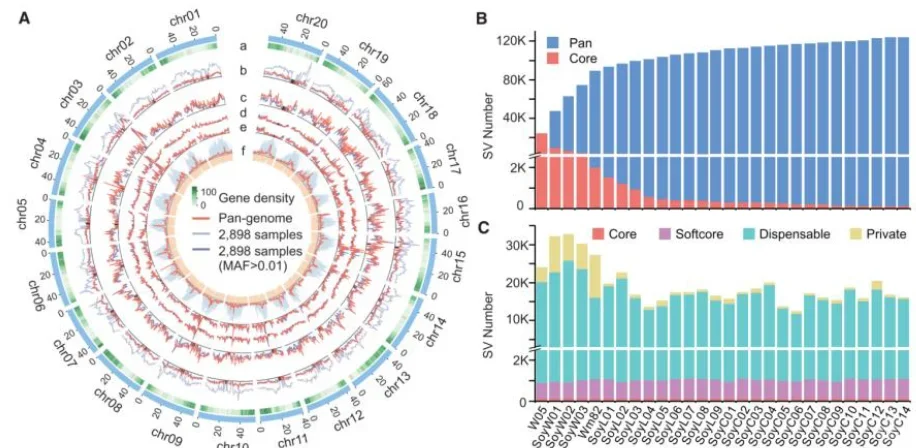
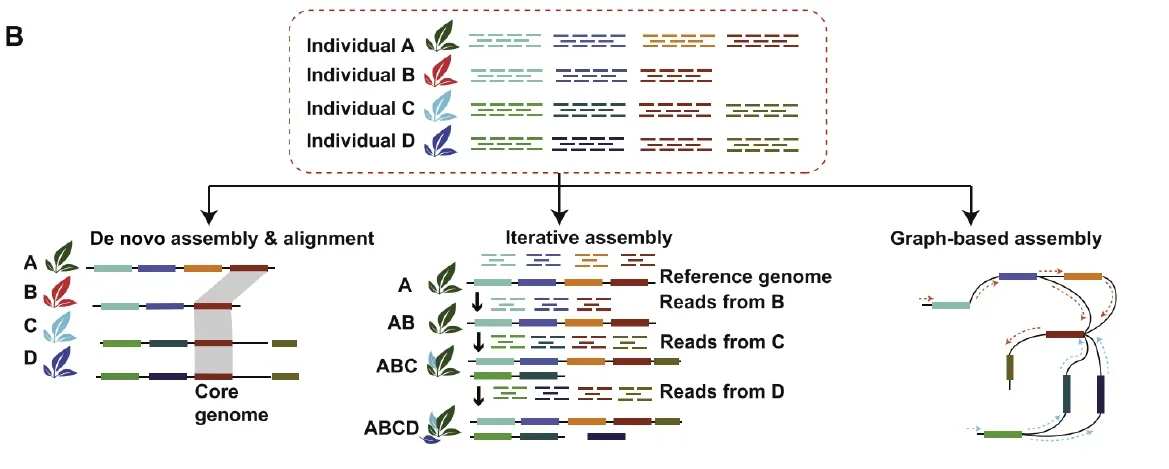
迭代和组装策略
迭代组装是基于参考基因组的泛基因组构建核心策略之一,其核心逻辑是从初始参考基因组出发,对样本进行逐个迭代处理:先将单个样本的测序数据比对到当前参考基因组,提取未比对(unmapped)的reads并组装为novel(新)序列,再将这些novel序列与当前参考基因组合并更新,形成新的参考基因组;随后用更新后的参考基因组处理下一个样本,重复“比对→提unmapped reads→组装→合并更新参考”的流程,直至所有样本处理完毕,最终得到包含物种所有可变序列的泛基因组。
优势 1. 逐步积累novel序列:每次迭代仅处理单个样本的unmapped reads,组装难度低,novel序列的准确性更高。 2. 减少冗余计算:更新后的参考基因组包含前序样本的特异序列,后续样本的未比对reads仅为自身特有序列,无需重复组装相似序列。 3. 适应性强:适用于多样本、中等测序深度的数据(如10-30×),尤其适合真菌、植物等基因组复杂度适中的物种(文档中以酵母为测试数据)。 4. 可追踪性:每个迭代步骤的novel序列可通过ID标记(如“r1.”“r2.”),便于后续追溯序列来源样本。
应用
◦ 物种:基因组大小适中(100Mb-1Gb)的物种。
◦ 数据类型:二代测序(Illumina)的双端reads,单样本测序深度≥10×(保证unmapped reads组装的覆盖度)。
◦ 研究目标:需构建高完整性泛基因组,同时区分不同样本特异序列的研究(如种群水平的基因可变分析)。
#数信院生信服务器 #生信 #生物信息学 #数据分析 #深度学习 #生物医学科研 #生信分析 #生信服务器 #生信入门 #图片来自于网络侵权可删
迭代组装是基于参考基因组的泛基因组构建核心策略之一,其核心逻辑是从初始参考基因组出发,对样本进行逐个迭代处理:先将单个样本的测序数据比对到当前参考基因组,提取未比对(unmapped)的reads并组装为novel(新)序列,再将这些novel序列与当前参考基因组合并更新,形成新的参考基因组;随后用更新后的参考基因组处理下一个样本,重复“比对→提unmapped reads→组装→合并更新参考”的流程,直至所有样本处理完毕,最终得到包含物种所有可变序列的泛基因组。
优势 1. 逐步积累novel序列:每次迭代仅处理单个样本的unmapped reads,组装难度低,novel序列的准确性更高。 2. 减少冗余计算:更新后的参考基因组包含前序样本的特异序列,后续样本的未比对reads仅为自身特有序列,无需重复组装相似序列。 3. 适应性强:适用于多样本、中等测序深度的数据(如10-30×),尤其适合真菌、植物等基因组复杂度适中的物种(文档中以酵母为测试数据)。 4. 可追踪性:每个迭代步骤的novel序列可通过ID标记(如“r1.”“r2.”),便于后续追溯序列来源样本。
应用
◦ 物种:基因组大小适中(100Mb-1Gb)的物种。
◦ 数据类型:二代测序(Illumina)的双端reads,单样本测序深度≥10×(保证unmapped reads组装的覆盖度)。
◦ 研究目标:需构建高完整性泛基因组,同时区分不同样本特异序列的研究(如种群水平的基因可变分析)。
#数信院生信服务器 #生信 #生物信息学 #数据分析 #深度学习 #生物医学科研 #生信分析 #生信服务器 #生信入门 #图片来自于网络侵权可删


