萨能奶山羊以产奶量高和适应性强著称,体尺性状是衡量其体型结构与生长发育的核心指标,与泌乳性能密切相关。解析体尺性状的遗传基础对奶山羊选育具有重要意义。然而,以往的全基因组关联分析(genome-wide association study,GWAS)主要聚焦于单核苷酸多态性(single-nucleotide polymorphism,SNP),忽略了基因组中广泛存在的结构变异(structural variant, SV)。将SV纳入GWAS分析,并结合多性状联合分析策略,有望挖掘出更多影响体型发育的关键遗传变异。
2026年2月5日,西北农林科技大学王喜宏课题组在《遗传》上发表了题为“整合结构变异的奶山羊体尺全基因组关联分析”的研究论文。该研究利用低深度全基因组测序及基因型填充技术,构建了高密度的SNP和SV数据集,创新性地整合了SV信息与多性状GWAS策略,系统解析了萨能奶山羊体尺性状的遗传机制。
研究人员以635只萨能奶山羊为研究对象,通过单性状(single-trait,ST)和多性状全基因组关联分析(multi-trait genome-wide association analysis,MT-GWAS),共鉴定出58个显著关联的SNP位点和5个显著关联的SV位点,主要位于TNFSF11、HDAC4、MURC等与骨骼及肌肉发育密切相关的基因上。值得注意的是,SNP MT-GWAS发现体长与十字部高在第10号染色体NRXN3基因附近存在重叠关联信号。进一步的共定位分析显示,共享因果变异模型的后验概率为0.988,证实了该区域存在影响多个体尺性状的共有遗传调控机制(如图所示)。此外,整合SV的分析还鉴定到了OXSM和FSIP2等新候选基因。
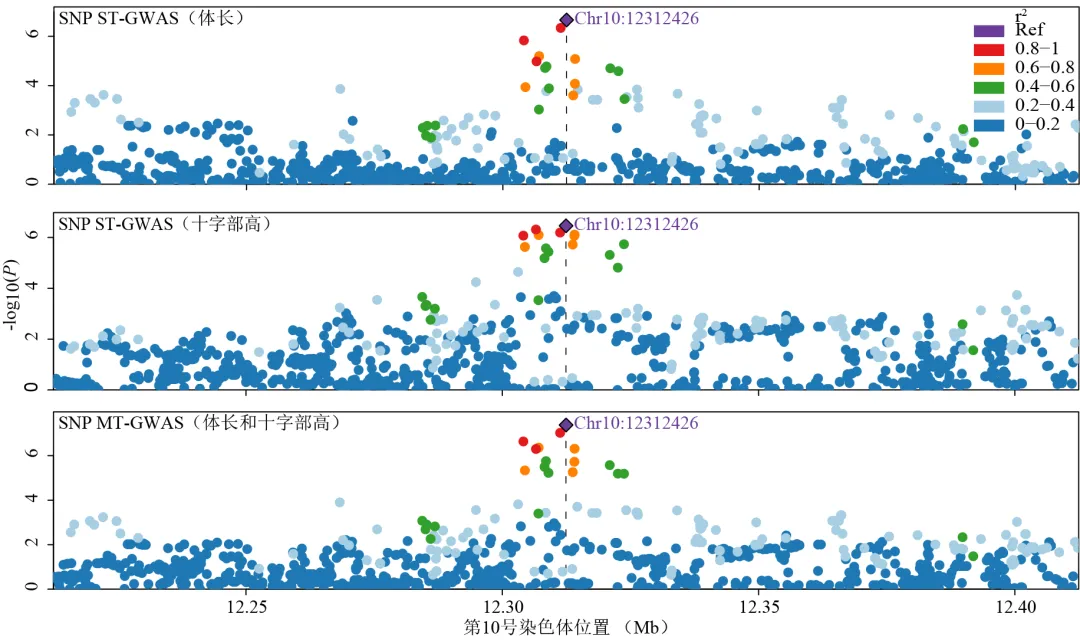
Chr10:12312426±100 kb区域的SNP GWAS局部关联图
综上所述,该研究表明整合SV的多性状GWAS策略能够鉴定到SNP之外的关联信号。研究发掘的候选基因及分子标记,丰富了萨能奶山羊体尺性状的遗传变异图谱,为开展分子标记辅助选择与体型精准选育提供了重要的理论依据。
西北农林科技大学王喜宏副教授为论文通讯作者,西北农林科技大学本科生冯衍帅为论文第一作者。该研究得到了国家自然科学基金项目和新疆维吾尔自治区重大科技专项的资助。
文章录用版链接:冯衍帅, 李嘉鑫, 赵炯尧, 曹家乐, 王兴权, 姚晓婷, 傅家琪, 王喜宏. 整合结构变异的奶山羊体尺全基因组关联分析. 遗传, 2026. DOI:10.16288/j.yczz.25-293.